少做实验or不做实验,短时间内发高分paper,也只有生物信息分析能做到了。小编在此分享一个家族基因生信分析套路,让你在短时间内喜提8分+SCI!

E2F转录因子(E2Fs)是一组编码转录因子家族的基因,参与多种肿瘤的发展。实验证据表明,E2Fs与乳腺癌的发生有关。然而,8种E2Fs的不同表达模式和预后价值尚未得到分析,因此作者将这8个基因于在线数据库进行进一步的分析验证。
1.在线分析网站
①Oncomine
https://www.oncomine.org/resource/login.html
②GEPIA
http://gepia.cancer-pku.cn/index.html
③Kaplan-Meier Plotter
http://kmplot.com/analysis/index.php?p=background
④cBioPortal
https://www.cbioportal.org/
⑤DAVID
https://david.ncifcrf.gov/summary.jsp
2.分析步骤
首先作者比较了E2Fs在癌症中的转录水平。利用Oncomine数据库,搜索E2F家族8个基因在各种癌中的转录水平,进行整理,宏观的展现出E2F家族在乳腺癌中表达的高低水平。
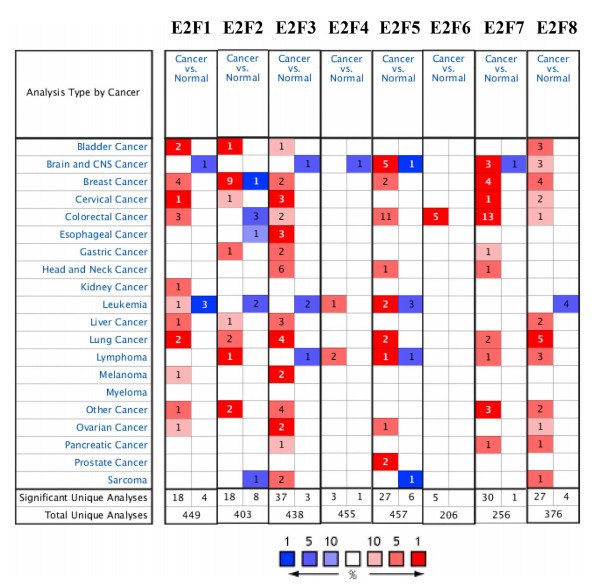
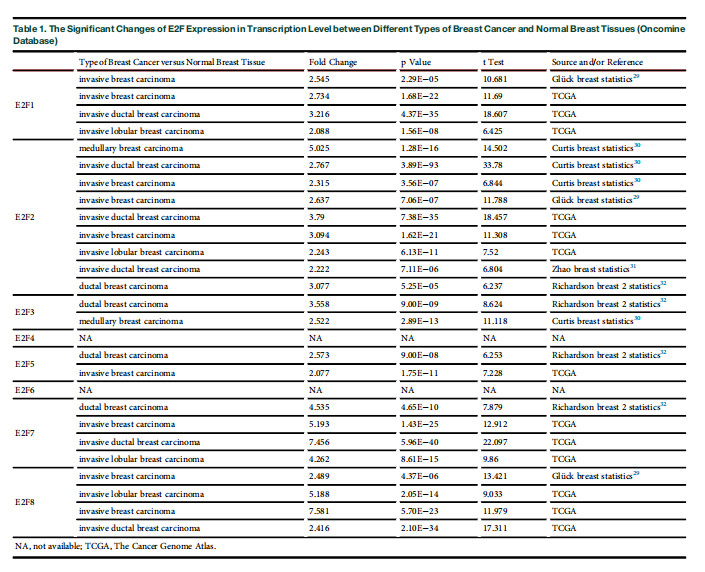
作者又在Oncomine 数据库中搜索并整理了不同类型乳腺癌与正常乳腺组织E2F转录水平的显着变化。
数据显示,乳腺癌组织中E2F1 mRNA表达水平显著升高。在Glück的数据集中,E2F1 在浸润性乳腺癌中高表达,与正常组织相比有2.545倍的改变。在癌症基因组图谱中,E2F1在浸润性中也过高表达。Glück等表明,浸润性乳腺癌中E2F2也较正常人增加(倍数变化=2.637)。
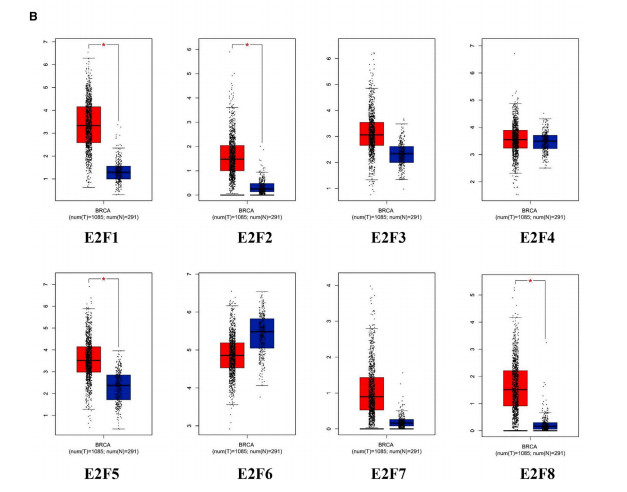
作者比较了正常组织和乳腺癌组织中E2F因子mRNA的表达。结果表明,E2F1、E2F2、E2F3、E2F5、E2F7和E2F8的表达水平不同。乳腺癌组织中E2F6的表达水平高于正常组织,而E2F6在乳腺癌组织中的表达水平低于正常组织。

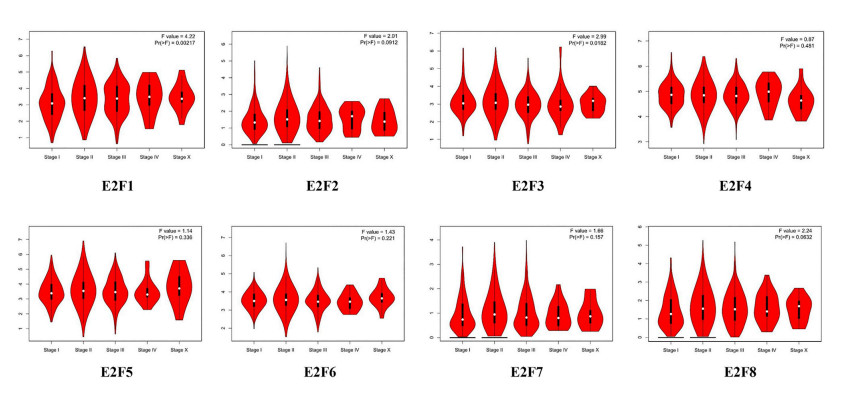
作者还分析了E2Fs在肿瘤中的表达。乳腺癌的分期。E2F1和E2F3组差异显著,而E2F2、E2F4、E2F5、E2F6、E2F7和E2F8组差异不显著。
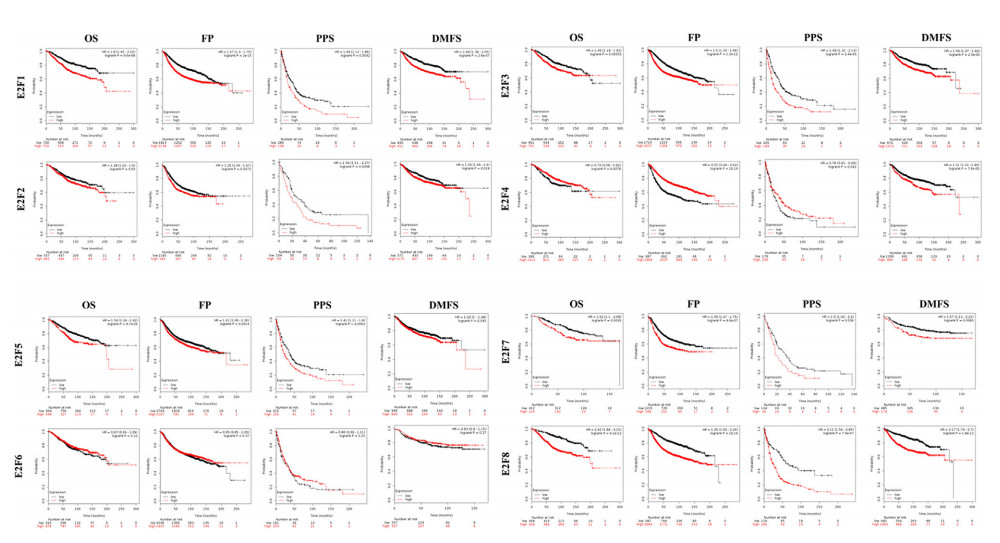
利用Kaplan-Meier Plotter在线网站分析E2Fs基因表达水平与乳腺癌患者生存率的关系。Kaplan-Meier曲线和log秩检验表明,E2f1-3、5、7和8的mRNA水平升高,而e2f4和6的mRNA水平显著降低。所有乳腺癌患者的总生存率(OS)、无进展生存(FP)和进展后生存(PPS)的ED(P<0.05)。乳腺癌患者e2f1-3、5、7和8的mRNA水平较高时,预测E2f4和6的mRNA水平较低,即OS、FP和PPS较高。
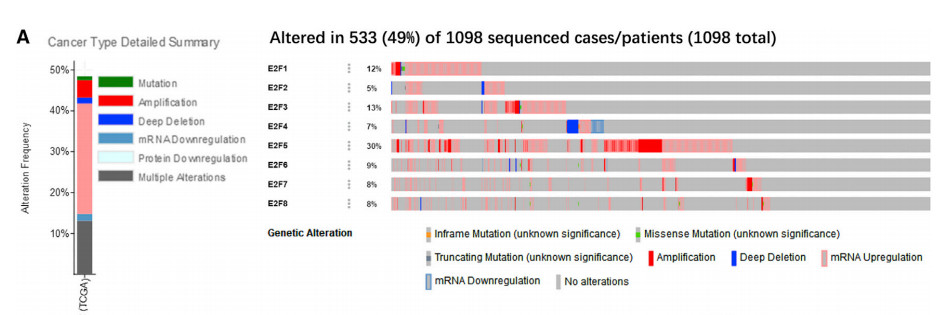
作者通过使用cBioPortal在线工具进行乳腺浸润性癌(癌症基因组图谱),分析了E2F的变化、相关性和网络分析。1,105例乳腺浸润性癌患者中,1,098份标本中E2FS发生改变(97%)。在样品的几乎2/3中检测到两种或多种变化(737个样本)。
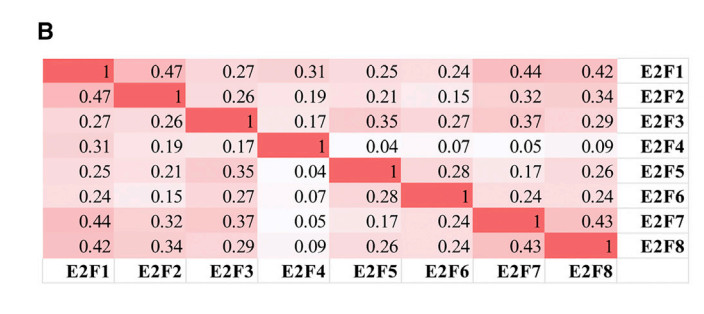
分析它们的mRNA表达通过CBioPortal在线工具来计算E2F彼此的相关性。结果表明:E2F1与E2F2、E2F4、E2F7和E2F8、E2F2与E2F1、E2F7和E2F8呈显著正相关;E2F2与E2F1、E2F7和E2F8呈显著正相关。
然后为E2fs和50个频繁的改变的基因进行网络互作用分析。结果表明,细胞周期相关基因CCND 1、CCNE 1、CCNE 2、CDC6、CDKN2A、RB1、RBL 1与E2F的改变密切相关。

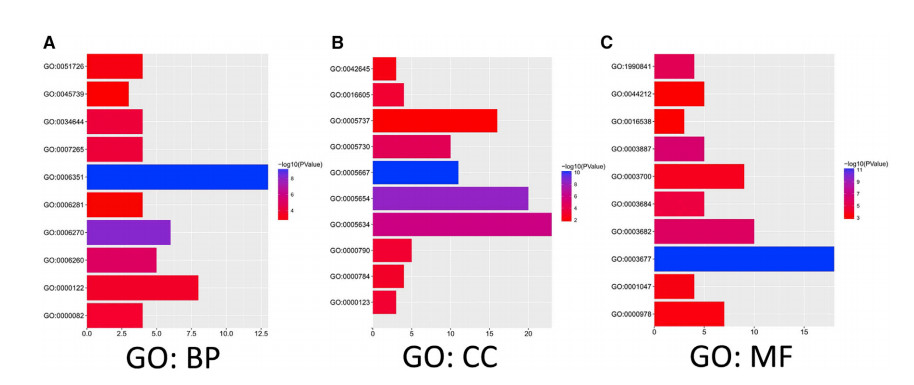
通过GO和KEGG分析,预测E2Fs的功能和与E2F改变密切相关的基因(DAVID在线网站)。
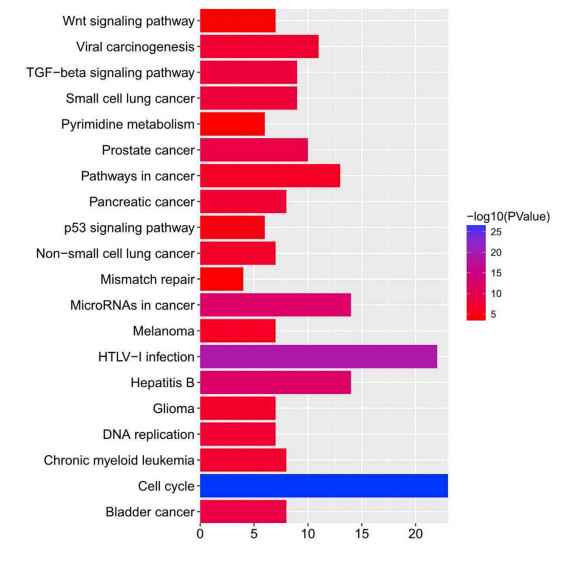
GO富集分析预测了目标宿主基因在生物过程、细胞组分和分子功能三个方面的功能作用,KEGG分析确定与E2F改变功能相关的通路和频繁改变的近邻基因。
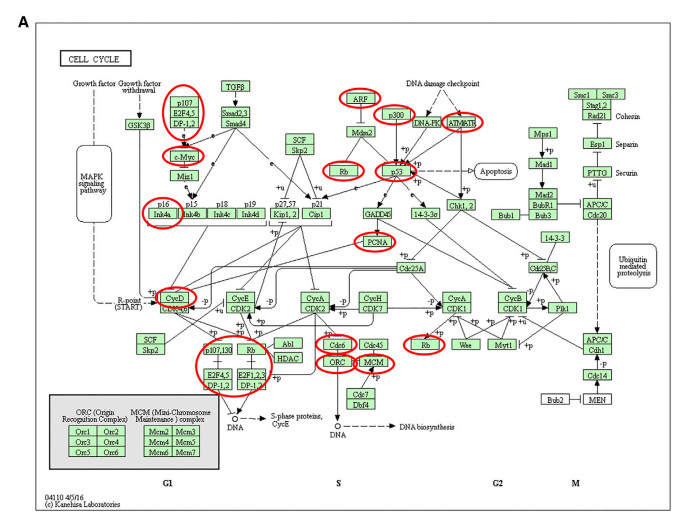
后作者展现了相关通路图,让结果一目了然。

一篇8分+的文章就这样轻松搞定,如果你已经了解了这个套路,可以给这个基因家族换个癌,或者按照这个套路换几个家族基因,发SCI是不是似乎并不难!